Desarrollan un nuevo método para detectar parentesco en datos de RNA
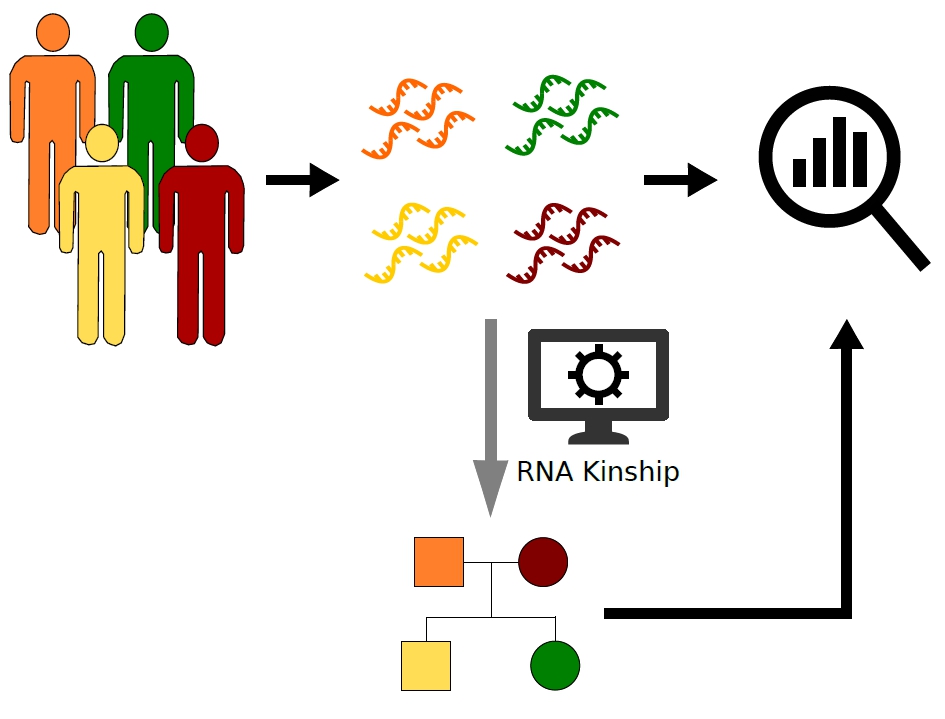
El grupo de investigación de Regulación Genómica del IGTP e IJC ha desarrollado un nuevo método para comprobar si las muestras de ARN secuenciado son de personas con un vínculo familiar. Han demostrado que se puede utilizar en grandes conjuntos de datos de secuencias de ARN para comprobar si las muestras provienen de individuos con un vínculo o no, o para reconstruir árboles genealógicos.
Los investigadores tienen acceso disponible a grandes cantidades de información científica en bases de datos, con información genética y otros datos asociados, de grandes grupos que hayan sido generados en estudios previos, sin la necesidad de generar esta información ellos mismos. Pero estos datos son anónimos, o se han anonimizado, lo que es un inconveniente para aquellos estudios en los que es importante conocer si hay relación de parentesco entre los individuos; en algunos casos, como el estudio de enfermedades hereditarias en familias, cuando tiene que haber relación, o en otros, cuando se estudia grandes muestras de población para encontrar patrones, en los que la existencia de vínculos familiares puede sesgar los estudios. Además, puede que se produzcan errores de etiquetaje durante el procesado de las muestras, ya que son procedimientos con una larga cadena de pasos, en los que intervienen muchas manos.
Es posible encontrar una relación de parentesco entre muestras individuales de datos genéticos (ADN). Sin embargo, muchos estudios solo generan información de ARN, una molécula similar generada en las células, pero que aporta información más específica sobre la salud de la célula y la importancia de las mutaciones de cada persona. "hasta el momento, los investigadores que trabajan con los datos de ARN tenían que tener acceso a la información de ADN para saber si las muestras eran de individuos con parentesco o no", explica Natalia Blay, la investigadora que ha llevado a cabo principalmente el estudio. "Era necesario descargar archivos enormes para poder llevar a cabo toda una nueva capa de análisis, y queríamos saber si podíamos obtener esta misma información de forma fiable directamente de los datos de la secuencia de ARN".
Después de desarrollar un método para detectar los individuos con parentesco a partir del ARN, el equipo lo probó contra un gran grupo de muestras del proyecto GCAT, de las cuales están perfectamente documentadas todas las relaciones familiares. Expertos del proyecto GCAT también han contribuido al estudio.
"Utilizando esta nueva técnica podemos detectar con éxito si las muestras de ARN son de personas con relación familiar y podemos, incluso, construir los árboles genealógicos", explica Tanya Vavouri, líder del estudio. "Los investigadores pueden establecer con total seguridad la relación familiar de los individuos en los muchos sets de datos disponibles de ARN secuenciado. Esto significa que se puede ahorrar tiempo y horas de computación para analizar los datos genómicos asociados que, por otro lado, no siempre están disponibles para todos los conjuntos de datos. En definitiva, la técnica permite utilizar la secuenciación de ARN de forma más fácil y económica a largo plazo".
El acceso a los datos y conocimientos del proyecto GCAT ha facilitado el desarrollo de esta nueva técnica. "Nuestro grupo tiene experiencia en el análisis de parentesco a partir del ADN", explica Rafa de Cid, director del proyecto GCAT. "Esto nos ha permitido colaborar con el equipo de Tanya Vavouri y desarrollar este método para ARN", añade.
La secuenciación de sets de ARN puede ser útil para el estudio de un gran número de enfermedades. Este tipo de estudio de datos es una fuente de información muy valiosa para aquellos estudios centrados en enfermedades particulares, su incidencia en la población o los factores de riesgo. Esta nueva metodología facilita el trabajo a científicos; está disponible gratuitamente online.
Article de referencia
Assessment of kinship detection using RNA-seq data. Natalia Blay, Eduard Casas, Iván Galván-Femenía, Jan Graffelman, Rafael de Cid, Tanya Vavouri, Nucleic Acids Research, gkz776,
Published: 10 September 2019
Financiación
Esta investigación ha recibido fondos del Ministerio de Economía y Competitividad (MICINN), [BFU2015-70581 and ADE 10/00026], la Agencia de Gestión de Ayudas Universitarias y de Investigación (AGAUR) [2017 SGR 1262 and 2017 SGR 529] y el programa CERCA de la Generalitat de Catalunya. Investigación al IJC ha recibido fondos de Obra Social Fundació "la Caixa", la Fundación Internacional Josep Carreras y Celgene España. La Agencia de Gestión de Ayudas Universitarias y de Investigación (AGAUR) ha financiado las becas de acceso abierto.
El Proyecto GCAT se ha podido realizado bajo el impulso del PMPPC-IGTP, en estrecha colaboración con el Banco de Sangre y Tejidos de Cataluña, y con total apoyo de la Dirección General de Investigación e Innovación, de la Consejería de Salud de la Generalidad de Cataluña.
Inicialmente, el proyecto GCAT se ha beneficiado del apoyo financiero del Ministerio de Salud, Servicios Sociales e Igualdad de España, y de la Consejería de Salud, de la Generalidad de Cataluña, a través de los fondos competitivos públicos del "Subprograma de acciones de dinamización del entorno investigador y tecnológico del SNS.2010 (ADE10 / 00026, FIS).

